B1.1.7 soy kümesi — Birleşik Krallık'ta ortaya çıkan SARS-CoV-2 soyunun yeni bir dizi başak mutasyonu ile tanımlanan ön genomik karakterizasyonu— @CovidGenomicsUK — COG-UK — SARSCOV2
İngiltere Başbakanı Boris JOHNSON, yeni koronavirüs varyantının %70 daha bulaşıcı olduğunu söyleyerek, Londra & ülkenin güneydoğusundaki vaka artışını buna bağladı.
Peki bu #SARSCoV2 türü daha bulaşıcı hale mi geldi? Ya da daha öldürücü? Geliştirilen aşılar etki etmeyecek mi?
Henüz bilmiyoruz.
Tek bildiğimiz #SARSCoV2 VUI-202012/01 (First Variant Under Investigation) varyantının İngiltere’ de hızlı bir şekilde yayıldığı.
Birleşik Krallık'ta ortaya çıkan SARS-CoV-2 soyunun yeni bir dizi başak mutasyonu ile tanımlanan ön genomik karakterizasyonu
Rapor yazan: Andrew Rambaut 1 , Nick Loman 2 , Oliver Pybus 3 , Wendy Barclay 4 , Jeff Barrett 5 , Alesandro Carabelli 6 , Tom Connor 7 , Tom Peacock 4 , David L Robertson 8 , Erik Volz 4 , COVID adına- 19 Genomics Consortium UK (CoG-UK) 9 .
- Edinburgh Üniversitesi
- Birmingham Üniversitesi
- Oxford Üniversitesi
- Imperial College London
- Wellcome Trust Sanger Enstitüsü
- Cambridge Üniversitesi
- Cardiff Üniversitesi
- MRC-University of Glasgow Virüs Araştırma Merkezi
- https://www.cogconsortium.uk 920
Özet
Son zamanlarda, COG-UK sürveyans veri setinde farklı bir filogenetik küme (soy B.1.1.7 olarak adlandırılır) tespit edildi. Bu küme, son 4 hafta içinde hızla büyüyor ve o zamandan beri diğer Birleşik Krallık lokasyonlarında da gözlemleniyor ve bu da daha fazla yayılmaya işaret ediyor.
- Mutasyon N501Y, reseptör bağlanma alanı (RBD) içindeki altı anahtar temas kalıntısından biridir ve insan ve murin ACE2'ye artan bağlanma afinitesi olarak tanımlanmıştır.
- Spike delesyonu 69-70del, insan immün tepkisinden kaçınma bağlamında tarif edilmiştir, ancak aynı zamanda diğer RBD değişiklikleriyle bağlantılı olarak birkaç kez meydana gelmiştir.
- P681H mutasyonu, biyolojik önemi olduğu bilinen bir konum olan furin klevaj bölgesine hemen bitişiktir.
Bu soyun hızlı büyümesi, dünya çapında gelişmiş genomik ve epidemiyolojik sürveyans ve antijenite ve enfektivite laboratuvar araştırmalarına duyulan ihtiyacı gösterir.
Arka fon
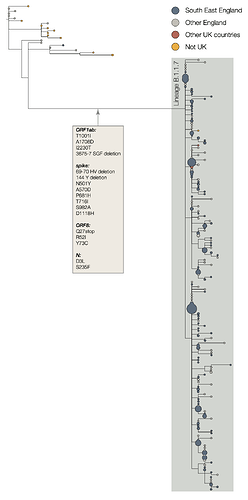
B.1.1.7 soyuna ait en eski örneklenmiş iki genom, 20-Eylül-2020'de Kent'te ve bir diğeri 21-Eylül-2020'de Greater London'dan toplandı. B.1.1.7 enfeksiyonları, Aralık 2020'nin başlarına kadar Birleşik Krallık'ta tespit edilmeye devam etmiştir. B.1.1.7 soyuna ait genomlar, çok sayıda soy tanımlayan mutasyonla iyi desteklenen bir monofiletik kuşak oluşturur (Şekil 1). 15 Aralık itibariyle, B.1.1.7 soyunda 1623 genom vardır. Bunlardan 519'u Greater London'da, 555'i Kent'te, 545'i hem İskoçya hem de Galler dahil olmak üzere Birleşik Krallık'ın diğer bölgelerinde ve 4'ü diğer ülkelerde örneklendi.
Şekil 1 | 30 Kasım-2020 tarihine kadar toplanan örnekler için B.1.1.7 soyunun filogenetik ağacı ve en yakın dış grup dizileri. Aynı konumdan ipuçları, alanı temsil edilen genomların sayısıyla orantılı olan daireler halinde daraltıldı. B.1.1.7 soyunda, her biri bir nükleotid değişikliği ile tanımlanan üç büyük alt kanat belirgindir. Bu sınıflardan biri, ORF8'de başka bir durdurma kodonu tarafından tanımlanır.
Soyları tanımlayan mutasyonlar ve evrim hızı
B.1.1.7 soyu, normalden daha fazla sayıda virüs genetik değişikliği taşır. Tespit edilmeden önce kökene özgü 14 amino asit değişiminin tahakkuku, COVID-19 pandemisine ilişkin küresel virüs genomik verilerinde bugüne kadar görülmemiş bir durumdur. SARS-CoV-2'nin küresel filogenetik ağacındaki çoğu dal, birkaç mutasyondan fazlasını göstermez ve mutasyonlar, zaman içinde nispeten tutarlı bir oranda birikir. Tahminler, dolaşımdaki SARS-CoV-2 soylarının, nükleotid mutasyonlarını ayda yaklaşık 1-2 mutasyon oranında biriktirdiğini göstermektedir (Duchene ve diğerleri, 2020).
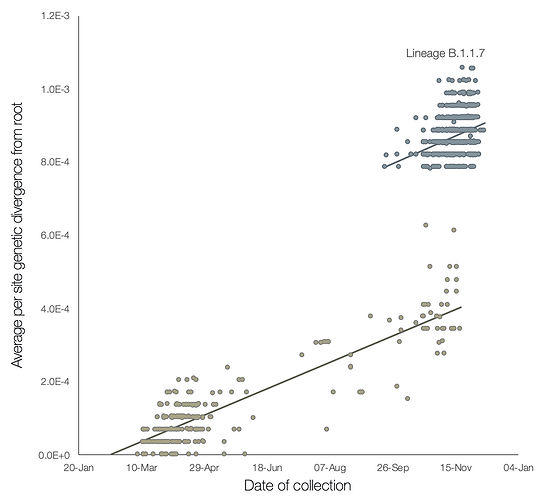
Bu gözlemlerin bir ön analizi, B.1.1.7 soyuna ve ilgili dış grup genomlarının bir seçimine yönelik genom örnekleme tarihine göre kökten uca genetik mesafelerin gerilemesini gösteren Şekil 2'de verilmektedir. Soy B.1.1.7 içindeki moleküler evrim hızı, diğer ilgili soylarınkine benzer. Bununla birlikte, B.1.1.7 soyu, pandeminin filogenetik kökünden daha farklıdır ve bu da, B.1.1.7'nin hemen atalarından olan filogenetik dalda daha yüksek bir moleküler evrim oranına işaret etmektedir. Ayrıca, bu dalda çıkarılan nükleotid değişiklikleri, ağırlıklı olarak amino asit değiştiricidir (14 eşanlamlı olmayan mutasyon ve 3 silme). Dalda 6 eşanlamlı mutasyon vardır. Bu, uyarlanabilir moleküler evrimi içeren bir süreci düşündürmektedir.
Şekil 2 | B.1.1.7 (mavi) soyuna ait diziler ve küresel filogenetik ağaçtaki (kahverengi) hemen dış grubundakiler için örnekleme tarihlerine göre kökten uca genetik mesafelerin gerilemesi. Regresyon çizgileri iki kümeye bağımsız olarak yerleştirilir. Regresyon gradyanı, dizi gelişim hızının bir tahminidir. Bu oranlar , B.1.1.7 ve dış grup veri setleri için sırasıyla 5.6E -4 ve 5.3E -4 nükleotid değişiklikleri / saha / yıl'dır.
Bu koşullar altında, hasta içi virüs popülasyonunun evrimsel dinamikleri ve üzerindeki seçici baskıların, tipik enfeksiyonda deneyimlenenlerden çok farklı olması beklenir. İlk olarak, bağışıklığı yetersiz / baskılanmış hastalarda doğal bağışıklık yanıtlarından seçim zayıf olacak veya olmayacaktır. İkinci olarak, antikor terapisinden kaynaklanan seçim, yüksek antikor konsantrasyonları nedeniyle güçlü olabilir. Üçüncüsü, eğer antikor tedavisi haftalarca kronik enfeksiyondan sonra uygulanırsa, virüs popülasyonu, antikor aracılı seçici basıncın uygulandığı sırada alışılmadık derecede büyük ve genetik olarak farklı olabilir, bu da çoklu virüs genetik değişikliklerinin doğrudan doğruya tespiti için uygun koşullar yaratır. seçim ve genetik otostop.
Bu düşünceler bizi, B.1.1.7 soyunun olağandışı genetik ayrışmasının, en azından kısmen, kronik olarak enfekte bir bireyle virüs evriminden kaynaklanmış olabileceği hipotezine götürür. Bu tür enfeksiyonlar nadir olmakla birlikte ve bunlardan ileriye doğru bulaşma muhtemelen daha da nadir olmakla birlikte, devam eden çok sayıda yeni enfeksiyon göz önüne alındığında olasılık dışı değildir.
Burada kronik enfeksiyonun B.1.1.7 varyantının kökeninde bir rol oynadığını tahmin etmemize rağmen, bu bir hipotez olarak kalır ve bu olayın kesin doğasını henüz çıkaramıyoruz.
Mutasyonların potansiyel biyolojik önemi
Tablo 1, B.1.1.7 kökene özgü eşanlamlı olmayan mutasyonlar ve silmelerin ayrıntılarını sağlar. Birçoğunun virüs spike proteininde meydana geldiğini not ediyoruz. Bunlar, reseptör bağlanma alanındaki (RBD) anahtar temas kalıntılarından biri olan başak pozisyonu 501'i içerir ve deneysel veriler, N501Y mutasyonunun ACE2 reseptör afinitesini artırabildiğini (Starr ve ark. 2020) ve P681H'yi, başakta S1 ve S2 arasında bir furin bölünme bölgesi oluşturur. SARS-CoV-2'nin S1 / S2 furin bölünme bölgesi yakından ilişkili koronavirüslerde bulunmaz ve solunum epitel hücrelerine girişi ve hayvan modellerinde bulaşmayı teşvik ettiği gösterilmiştir (Hoffmann, Kleine-Weber ve Pöhlmann 2020; Peacock et al. . 2020; Zhu ve diğerleri 2020). N501Y, bir fare modelinde artmış enfektivite ve virülans ile ilişkilendirilmiştir (Gu ve ark. 2020).
Aynı zamanda 69-70 bölgelerindeki iki amino asidin delesyonu da mevcuttur - bu mutasyon, Spike'ın N terminal alanında (McCarthy ve diğerleri 2020; Kemp ve diğerleri 2020) gözlemlenen bir dizi tekrarlayan delesyondan biridir ve çeşitli RBD mutasyonlarına bağlı birçok soyda görülmüştür. Örneğin, Danimarka'da Y453F RBD mutasyonunun arka planında vizonla ilişkili salgında ve N439K RBD mutasyonu ile bağlantılı olarak insanlarda ortaya çıktı ve küresel genom verilerinde nispeten yüksek frekansını (~ 3000 dizi) hesaba kattı.
Tablo 1 | Eşanlamlı olmayan mutasyonlar ve silmelerin, B.1.1.7 soyuna yol açan dalda meydana geldiği anlaşılır.
| gen | nükleotid | amino asit |
|---|---|---|
| ORF1ab | C3267T | T1001I |
| C5388A | A1708D | |
| T6954C | I2230T | |
| 11288-11296 silme | SGF 3675-3677 silme | |
| sivri uç | 21765-21770 silme | HV 69-70 silme |
| 21991-21993 silme | Y144 silme | |
| A23063T | N501Y | |
| C23271A | A570D | |
| C23604A | P681H | |
| C23709T | T716I | |
| T24506G | S982A | |
| G24914C | D1118H | |
| Orf8 | C27972T | Q27stop |
| G28048T | R52I | |
| A28111G | Y73C | |
| N | 28280 GAT-> CTA | D3L |
| C28977T | S235F |
Ani yükselmenin dışında, ORF8 Q27stop mutasyonu ORF8 proteinini keser veya onu inaktif hale getirir ve böylece daha fazla aşağı yönde mutasyonların oluşmasına izin verir. Pandemi sırasında erken dönemde ORF8 ekspresyonunun kaybına yol açan delesyonlara sahip çoklu virüs izolatları dünya çapında izole edildi; Singapur'da hem kesilmiş bir Orf7b hem de kesilmiş ORF8 ekspresyonuna yol açan bir delesyonla büyük bir küme dahil. 382nt'lik bir delesyona sahip olan Singapurlu suş, daha hafif bir klinik enfeksiyon ve daha az enfeksiyon sonrası enflamasyonla ilişkilendirildi, ancak bu küme, Singapur'un başarılı bir şekilde kontrol önlemlerini uyguladıktan sonra Mart ayı sonunda yok oldu (Young ve ark. 2020). Daha sonraki çalışma, ORF8 delesyonunun, insan birincil hava yolu hücrelerinde virüs replikasyonu üzerinde, silinmeyen virüslere kıyasla çok ılımlı bir etkiye sahip olduğunu bulmuştur. silme ile virüslere kıyasla hafif bir replikasyon gecikmesine yol açar (Gamage ve diğerleri 2020). ORF8 genellikle 121 amino asit uzunluğunda olduğundan, B.1.1.7 soyunda gözlenen 27 konumundaki durdurma kodonu büyük olasılıkla işlev kaybına yol açar.
Son olarak, ORF1ab'de (C913T, C5986T, C14676T, C15279T, C16176T) ve M geninde (T26801C) 5 ile 6 eşanlamlı mutasyon vardır.
Sonuç
Birleşik Krallık'ta, reseptör bağlanma alanı dahil olmak üzere beklenmedik şekilde çok sayıda genetik değişiklikle ilişkili ve furin bölünme bölgesi ile ilişkili hızla büyüyen bir soy bildiriyoruz. (İ) bu mutasyonlardan bazılarının deneysel olarak tahmin edilen ve makul fenotipik sonuçları, (ii) kombinasyon halinde mevcut olduklarında bilinmeyen etkileri ve (iii) Birleşik Krallık'ta B.1.1.7'nin yüksek büyüme oranı göz önüne alındığında, bu yeni soy acil laboratuvar karakterizasyonu ve dünya çapında gelişmiş genomik sürveyans gerektirir.
Referanslar
Avanzato, Victoria A., M. Jeremiah Matson, Stephanie N. Seifert, Rhys Pryce, Brandi N. Williamson, Sarah L. Anzick, Kent Barbian, vd. 2020. "Örnek Olay: Kanserli Asemptomatik Bağışıklığı Bozulmuş Bir Kişiden Uzun Süreli Bulaşıcı SARS-CoV-2 Dökülmesi." Cell, Kasım. https://doi.org/10.1016/j.cell.2020.10.049 224.
Choi, Bina, Manish C. Choudhary, James Regan, Jeffrey A. Sparks, Robert F. Padera, Xueting Qiu, Isaac H. Solomon, vd. 2020. "Bağışıklığı Bozulmuş Bir Konakta SARS-CoV-2'nin Kalıcılığı ve Gelişimi." New England Journal of Medicine 383 (23): 2291–93.
Duchene, Sebastian, Leo Featherstone, Melina Haritopoulou-Sinanidou, Andrew Rambaut, Philippe Lemey ve Guy Baele. 2020. "Zamansal Sinyal ve SARS-CoV-2'nin Filodinamik Eşiği." Virus Evolution 6 (2): veaa061.
Young, Barnaby E. vd. 2020. "SARS-CoV-2 Genomundaki Büyük Bir Delesyonun Enfeksiyonun Şiddeti ve İnflamatuar Yanıt Üzerindeki Etkileri: Bir Gözlemsel Kohort Çalışması." 2020. Lancet 396 (10251): 603–11.
Gamage, Akshamal M., Kai Sen Tan, Wharton OY Chan, Jing Liu, Chee Wah Tan, Yew Kwang Ong, Mark Thong ve diğerleri. 2020. "İnsan Nazal Epitel Hücrelerinin SARS-CoV-2 ve ORF8 Eksik 382-Nt Delesyon İzolatı ile Enfeksiyonu Benzer Viral Kinetik ve Konak Transkripsiyon Profillerini Ortaya Çıkarıyor." PLoS Patojenleri 16 (12): e1009130.
Gu, Hongjing, Qi Chen, Guan Yang, Lei He, Hang Fan, Yong-Qiang Deng, Yanxiao Wang ve diğerleri. 2020. "BALB / c Farelerde SARS-CoV-2'nin Aşı Etkinliğini Test Etmek İçin Uyarlanması." Science 369 (6511): 1603–7.
Hoffmann, Markus, Hannah Kleine-Weber ve Stefan Pöhlmann. 2020. "SARS-CoV-2'nin Başak Proteinindeki Çok Bazlı Bir Bölünme Bölgesi, İnsan Akciğer Hücrelerinin Enfeksiyonu İçin Gereklidir." Molecular Cell 78 (4): 779–84. E5.
Kemp, SA, DA Collier, R. Datir, S. Gayed, A. Jahun, M. Hosmillo, Iatm Ferreira, vd. 2020. "Nötralize Edici Antikorlar, Spike Aracılı SARS-CoV-2 Kaçınmasını Sağlar." Bulaşıcı Hastalıklar (HIV / AIDS hariç). medRxiv. https://doi.org/10.1101/2020.12.05.20241927 109
McCarthy, Kevin R., Linda J. Rennick, Sham Nambulli, Lindsey R. Robinson-McCarthy, William G. Bain, Ghady Haidar ve W. Paul Duprex. 2020. "SARS-CoV-2 Spike Glycoprotein Drive Antikor Kaçışındaki Doğal Delesyonlar." Mikrobiyoloji. bioRxiv.
Peacock, Thomas P., Daniel H. Goldhill, Jie Zhou, Laury Baillon, Rebecca Frize, Olivia C. Swann, Ruthiran Kugathasan, vd. 2020. "SARS-CoV-2 Spike Proteininin Furin Bölünme Bölgesi, Hava Yolu Hücrelerinde Gelişmiş Replikasyon nedeniyle İletim için Anahtar Belirleyicidir." Cold Spring Harbor Laboratuvarı. https://doi.org/10.1101/2020.09.30.318311 58.
Starr, Tyler N., Allison J. Greaney, Sarah K. Hilton, Daniel Ellis, Katharine HD Crawford, Adam S. Dingens, Mary Jane Navarro, vd. 2020. "SARS-CoV-2 Reseptör Bağlama Alanının Derin Mutasyonel Taraması, Katlama ve ACE2 Bağlamasındaki Kısıtlamaları Ortaya Çıkarıyor." Celi 182 (5): 1295–1310.e20.
Zhu, Yunkai, Fei Feng, Gaowei Hu, Yuyan Wang, Yin Yu, Yuanfei Zhu, Wei Xu, ve diğerleri. 2020. "SARS-CoV-2 Spike Proteininin S1 / S2 Sınırı, Hücre Giriş Yollarını ve İletimi Modüle Ediyor." Cold Spring Harbor Laboratuvarı. https://doi.org/10.1101/2020.08.25.266775 80.
 Reviewed by ümitse
on
22:40:00
Rating:
Reviewed by ümitse
on
22:40:00
Rating:












Hiç yorum yok: